obiecte
abstract
În ultimii 30 de ani, am efectuat multe studii de bază pe două Oryza sativa subsp. indica, Zhenshan 97 (ZS97) și Minghui 63 (MH63). Pentru a îmbunătăți rezoluția multora dintre aceste studii, am creat două ansambluri de referință ale genomului de referință folosind tehnologii de secvențiere de ultimă generație. Folosind tehnologia PacBio SMRT, am produs mai mult de 108 (ZS97) și 174 (MH63) Gb date secvență brută din 166 (ZS97) și 209 (MH63) grupuri de clone BAC și am generat
74 (MH63) Gb întreg genom pereche secvență finală secvență (WGS) secvență date cu tehnologie de secvențiere Illumina. Cu aceste date, am compilat cu succes două standarde standard de platină care au fost publicate. Iată seturile complete de date brute utilizate pentru a genera aceste două ansambluri genomice de referință. Aceste seturi de date pot fi utilizate pentru a testa noi programe pentru o mai bună asamblare și adnotare a genomului, pentru a ajuta la descoperirea de noi perspective asupra structurii, funcției și evoluției genomului și pentru a oferi sprijinul necesar pentru cercetarea biologică în general.
Rezumatul metadatelor
Descărcați fișierul de metadate
Fișier de metadate accesibil la mașină care descrie datele raportate (format card ISA)
Context și rezumat
metode
Construcția bibliotecii BAC și secvențierea secvențială
Cele două biblioteci BAC utilizate în acest studiu au fost publicate anterior 11. Pe scurt, ADN-ul genomic parțial digerat (adică Hin dIII) și selectat în funcție de mărime din fiecare soi a fost donat în situsul Hin dIII al pAGIBAC1 și transformat în celule competente Escherichia coli DH10B T1. Ambele biblioteci, denumite OSIZBa (ZS97) și OSIABa (MH63), conțineau 36.864 de clone BAC, dimensiunile medii ale inserției erau
125 kb (MH63) și au fost acoperite
10, 7 × (MH63) din fiecare genom 11. În plus, au fost generate 33, 969 (ZS97) și 35, 549 (MH63) secvențe bidirecționale de capăt BAC (BES) pentru prima jumătate a fiecărei biblioteci 11.
Hărți fizice
Hărțile fizice cu acoperire redusă (PM) descrise mai sus folosind metoda de amprentă digitală SNaPshot au fost descrise mai sus11. Am convertit cei doi PM folosind metoda KeyGene Whole Genome Profiling (WGP) 12. PM-urile WGP FingerPrint Contig (FPC) au fost construite în patru etape: (1) Pregătirea ADN-ului BAC, (2) pregătirea plasmidelor WGP BAC cu adaptoare de indexare și secvențiere, (3) secvențierea Illumina și (4) prelucrarea bioinformatică. La pasul 4, folosind scripturi de decodare WGP, 99.996 (ZS97) și 103.609 (MH63) au fost deconvoluate etichete WGP unice și s-au marcat 32.829 (89,1%) și 30.749 (89,3%) BAC-uri în bibliotecile ZS97 și MH63, resp. Folosind etichete de secvență WGP pentru fiecare clonă BAC din fiecare bibliotecă, au fost construite două PM noi cu software-ul FPC 13 (versiunea 9.4). După editarea manuală și integrarea cu PM-urile SNaPshot anterioare, PM-urile îmbunătățite ZS97 și MH63 au fost formate din 539 și 401 contigs, conținând 28, 372 și 24, 519 clone și 4, 457 și 6, 230 clone ca singletoni. Dimensiunile contigului total au fost estimate la 342 Mb pentru ZS97 (N50 = 940 kb) și 349 Mb pentru MH63 (N50 = 1.270 kb).
Secvențierea clonei PacBio BAC
Clonele BAC Minime Paving Path (MTP) din fiecare PM au fost selectate automat folosind un script personalizat și aranjate manual în farfurii de bibliotecă MTP etichetate OSIZBzz (ZS97) și OSIABzz (MH63) și stocate la -80 ° C. Un total de 4, 714 și Au fost colectate 4.751 de clone BAC MTP pentru ZS97 și MH63. Listele complete ale clonelor MTP sunt disponibile în tabelul suplimentar 1a-b.
Pentru secvențierea clonei PacBio BAC, clonele MTP BAC au fost inoculate în blocuri de creștere cu 96 de godeuri adânci, crescute peste noapte la 37 ° C, centrifugate pentru a peleta celulele și apoi stocate la -80 ° C până la utilizare. BAC-urile au fost apoi formate prin fuzionarea puțurilor din blocuri înghețate într-una din cele șase configurații: adică asociații de nave individuale (12 BAC pe piscină), două asociații de rânduri (24 BAC pe piscină), patru grupuri de coloane (32 BAC pe piscină), 6 grupuri (48 BAC per pool), opt asociații de coloane (64 BAC per pool) sau grupuri de pensiune completă (96 BAC per pool). ADN-ul a fost apoi extras din fiecare amestec folosind un protocol standard de izolare a plasmidei plasmidei ADN 14 de liză alcalină. Un total de 166 (ZS97) și 209 (MH63) pool-uri au fost secvențiate (vezi schema noastră de pooling detaliată în tabelul suplimentar 2a-b). Folosind 16 μg de ADN plasmidic grupat, bibliotecile de secvențe PacBio au fost preparate în conformitate cu următoarele protocoale ale producătorului, așa cum sunt descrise pentru prepararea șablonului Blue Pippin de 20 kb. Secvențierea SMRT a fost efectuată pe un instrument PacBio RSII folosind chimia secvenței P5/C3 și filme de 3 h.
Producție de lectură brută cu PacBio
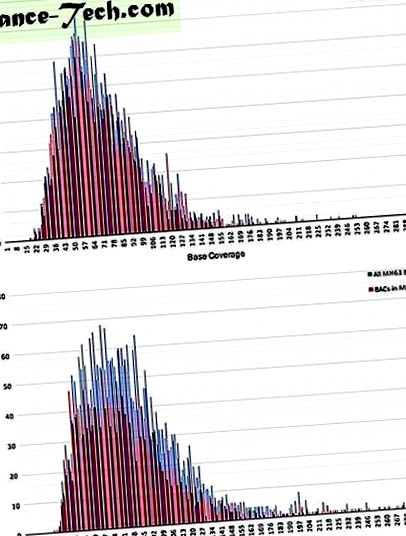
Analizele de subcaten pentru ambele grupuri de secvențe ZS97 și MH63 BAC au fost efectuate folosind Portalul PacBio SMRT (versiunea 2.3.0). Pentru ZS97, datele de la 227 celule SMRT (care numără reacțiile de refacere) au fost separate și filtrate (adică, folosind protocolul RS_Subreads, lungimea minimă de citire a polimerazei = 50 bp, calitatea minimă de citire a polimerazei = 75 și lungimea minimă a subcitirii = 50 bp), care este rezultatul unui total de 107,5 Gb de date de secvență utilizabile (numărul total de reacții ale polimerazei = 11,6 M, numărul de polimeraze N50 = 12,8 kb; numărul total de scufundări = 17,7 M, lungimea medie subcutanată = 5,7 kb, subcitirea N50 = 8,0 kb). Pentru MH63, datele de la 317 celule SMRT au fost procesate ca mai sus (174 Gb de date utilizabile; 18,2 M polimerază citită, polimerază citită N50 = 12,1 kb; 26,8 M subcitiri, lungime medie a subcitirii = 5,5 kb, subcitită N50 = 7, 8 kb).
Compilarea datelor PacBio și identificarea secvenței BAC
Imagine la dimensiune completă
Material vegetal, construirea unei biblioteci ADN pentru secvențierea iluminării
De asemenea, am folosit tehnologia de secvențiere a citirii scurte a lui Illumin pentru a secvența genomurile ZS97 și MH63 folosind abordarea pistolului cu întregul genom (WGS). Materialele vegetale au fost cultivate într-o seră și frunzele vechi de 4 săptămâni au fost folosite pentru a extrage ADN genomic folosind proceduri standard. Biblioteci pereche, inclusiv inserții mici (
300 bp) și două biblioteci de inserții mari (5 kb, 10 kb) au fost pregătite cu seturi de capete și perechi de perechi Illumina (Tabelul 1). Cel puțin 5 μg de ADN genomic au fost fragmentate prin nebulizare cu azot gazos comprimat pentru biblioteci de inserție de inserție scurtă. O cantitate mai mare de ADN genomic de înaltă calitate (10-30 μg) a fost necesară pentru a construi o bibliotecă cu o inserție lungă de pereche mate. Bibliotecile de secvențe Illumina au fost pregătite conform protocolului producătorului. Bibliotecile au fost secvențiate pe Illumina HiSeq 2000 conform protocoalelor standard Ilulina (Illumina, San Diego, CA). Cantitatea totală de date secvenței brute generate pentru fiecare soi a fost
97, 5 Gb de date pentru ZS97 și
Date de 74,0 Gb pentru MH63. După o serie de pași de filtrare a datelor, acestea au fost utilizate pentru a elimina citirile artificiale cauzate de duplicarea PCR și contaminarea adaptorului, pentru un total de - 87, 4 Gb de citire de înaltă calitate (> 200 ×) pentru ZS97 și
Au fost obținute 67,9 Gb (> 170 ×) pentru MH63 (Tabelul 1). Calitatea bibliotecii a fost verificată prin determinarea distribuției dimensiunilor inserției și adâncimii secvenței. Lungimile reale de inserare au fost determinate prin maparea capetelor împerecheate la O. sativa subsp. japonica cv. Genomul de referință Nipponbare (Nipponbare RefSeq) 17 .
Tabel în dimensiune completă
Illumina citește pre-procesarea și asamblarea de novo
Am folosit o nouă abordare hibridă care combină asamblarea de novo și metodele de referință 18 pentru a compila citirile Illumina pentru fiecare genom. Toate citirile secvențiate din ZS97 și MH63 au fost corectate cu Medusa 19 și Quake 20. Citirile corectate au fost trunchiate la capetele lor dacă calitatea secvenței lor a fost mai mică de 20 folosind fastx_tool_kit (//hannonlab.cshl.edu/fastx_toolkit/index.html), iar contoare au fost îndepărtate folosind cutadpat 21 (//github.com/) Marcely/cutadapt /). Datele procesate au fost apoi mapate la un Nipponbare RefSeq folosind un BWA 22. Toate citirile care au fost mapate la zona continuă au fost luate individual și zona de acoperire contiguă a fost definită ca un bloc. Definiția blocurilor și superblocurilor este aceeași cu cea descrisă mai sus 23, cu excepția faptului că lungimea noastră minimă a superblocului a fost de 20 kb și suprapunerea superblocului a fost de 2 kb. La nivel local, am compilat de novo toate citirile colectate în superbloc folosind SOAPdenovo 24. Au fost testate o serie de k-meri diferiți și au fost reținute contigurile rămase cu cele mai mari valori N50. Contigurile rezultate au fost asamblate cu AMOS 25 folosind cromozomii lor de referință ca ghiduri. Citirile neasociate au fost mapate la genomul indica 9311 și asamblate folosind același procedeu.
Nucmer 27 a fost apoi folosit pentru a alinia toate supercontrolele la Nipponbare RefSeq. Am verificat apoi adâncimea de acoperire și cartografierea golurilor dintre zonele adiacente. Citirile cartografiate au fost selectate prin extinderea golurilor în podurile genomului MH63 și ZS97 cu 200 bp pe ambele părți ale fiecărui gol. Toate numerele selectate în aceste zone au fost compilate folosind cap3 28. Contigurile asamblate au fost aliniate cu două supercontrole continue și conectate pe baza aranjamentului lor secvențial.
Pentru a încorpora secvențe specifice MH63 și ZS97 care nu erau prezente în genomurile Nipponbare și 9311, am efectuat o asamblare completă a genomului de novo utilizând toate citirile SOAPden 24 prelucrate și apoi am aliniat schelele de novo asamblate cu superconductele combinate și am legat în continuare supercontrolurile corespunzătoare . În cele din urmă, schela a fost efectuată folosind SSPACE 29 și golurile au fost umplute cu gapCloser (//sourceforge.net/projects/soapdenovo2/files/GapCloser/).
Statisticile finale ale kiturilor WGS Illumina ZS97 și MH63 sunt prezentate în tabelele 2 și 3 și au fost utilizate pentru a umple golurile dintre contigurile PacBio adiacente.
Tabel în dimensiune completă
Tabel în dimensiune completă
Construirea pseudomoleculelor în doi pași
În primul pas, toate datele secvenței BAC au fost inserate în Genome Puzzle Master 16 (GPM) pentru a crea contigs de secvență bazate pe PacBio folosind hărți fizice și manuale de referință Nipponbare RefSeq 17. GPM este o conductă semi-automată care a fost dezvoltată pentru a integra datele relației logice (de exemplu, hărți fizice) în secvențe de schele în secvențe de interval cromozomial. Ca rezultat, 318 (ZS97) și 216 (MH63) contigs asamblate au fost aranjate și orientate, precum și ancorate la cromozomii lor respectivi, după inspecția manuală, editarea și îndepărtarea excesului. Ansamblurile finale bazate pe PacBio au constat din secvențe din 3862 (ZS97, inclusiv 57 non-MTP) și 3254 (MH63, inclusiv 77 non-MTP) BAC-uri unice.
Deoarece am folosit o strategie de secvențiere bazată pe hărți fizice în acest studiu, lacunele din hărțile noastre fizice WGP sunt principalele cauze ale pauzelor în ansamblurile bazate pe PacBio. Prin urmare, al doilea pas a fost de a umple golurile prin integrarea datelor ansamblului Illumina WGS. Cu toate acestea, pentru a minimiza impactul datelor potențial incorecte în datele Illumina, am folosit doar contra Illumina, care ar putea conecta complet două contacte PacBio adiacente în acest pas. Un total de 81 de goluri în ZS97 (8.988.328 bp) și 35 în MH63 (3.127.191 bp) au fost umplute cu 76 (ZS97) și 35 (MH63) secvențe de iluminare contină (Tabel suplimentar 4a-b). Ca rezultat, pseudomoleculele hibride finale (ZS97RS1 și MH63RS1) conțineau 237 (inclusiv 2 necluse) și 181 (inclusiv 2 neconfirmate) contigs pentru ZS97 și MH63.
Înregistrări de date
Toate datele non-secvențiale sunt disponibile pe iPlant Datastore (Tabelul 4). Bibliotecile OSIZBa și OSIABa BAC, sau clonele individuale BAC, pot fi obținute de la Centrul de resurse BAC/EST al AGI la www.genome.arizona.edu/orders. Secvențele finale BAC au fost stocate anterior în GenBank sub numerele de acces KG737749 - KG771717 (ZS97, citarea datelor 1: GenBank KG737749 - KG771717) și KG702200 - KG737748 (MH63, citarea datelor 2: GenBank KG702200 - KG737748).
Tabel în dimensiune completă
Datele brute ale secvenței PacBio sunt disponibile în NCBI Short Read Archive (SRA) sub numerele de acces SRP071597 (ZS97, Data Citation 3: NCBI Sequence Read Archive SRP071597) și SRP071598 (MH63, Data Citation 4: NCBI Sequence Read Archive SRP071598). Toate datele secvenței Illumina pot fi găsite sub numărul de acces SRP071944 (ZS97 și MH63, Data Citation 5: NCBI Sequence Read Archive SRP071944). Din cauza unei erori neașteptate pe disc, am pierdut datele brute ale secvenței PacBio din 57 de pool-uri. Din fericire, toate rulările HGAP au fost arhivate în iPlant Datastore sub „Death-Jobs”, unde a fost posibil să se obțină substraturi filtrate ale acestor grupuri corespunzătoare. Datele compilate de la Illumina sunt disponibile pe ansamblul NCBI sub numerele de acces GCA_001618795 (ZS97, citarea datelor 6: ansamblul NCBI GCA_001618795) și GCA_001618785 (MH63, citarea datelor 7: ansamblul NCBI GCA_001618785).
Pseudomoleculele genomului final (versiunea 1) pentru fiecare genom de referință au fost stocate în ansamblul NCBI sub numerele de acces GCA_001623345 (ZS97RS1, citarea datelor 8: ansamblul NCBI GCA_001623345) și GCA_001623365 (MH63RS1, citarea datelor 9: ansamblul NCBI_G0065).
Verificarea tehnică
În esență, fiecare bibliotecă BAC echivalentă genomică a fost proaspăt cultivată în seturi copiate de plăci cu 384 de godeuri și s-a efectuat unirea tridimensională pe celule bacteriene, urmată de creșterea celulară și extracția ADN-ului plasmidic utilizând chimia lizei alcaline. Ansamblurile ADN au fost digerate cu enzime de restricție (EcoRI/MseI), urmate de ligarea oligomerilor de dimensiuni de piscină care au fost proiectate pentru a localiza în mod specific adresele clonelor BAC și a se asocia cu secvențele. După amplificarea moleculelor mixte, s-a efectuat secvențierea Illumina și datele rezultate au fost analizate pentru a identifica o etichetă de secvență de 50 bp pentru fiecare adresă specifică a clonei BAC și pentru a genera seturi de benzi ca intrare în FPC. FPC a fost rulat sub setări de severitate ridicată (HS): mai întâi cu „toleranță = 0 [fix], Cutoff = 1e-15”, apoi selectând DQ (în 3 pași: Cutoff = 1e-18, 1e-21, 1e -24 ) a fost folosit pentru a distribui contiguri problematice. După crearea PM-urilor HS rezultate, am efectuat pasul de îmbinare Ends-to-Ends (Cutoff = 1e-9) și am încorporat singletonii marcați în contigs (Cutoff = 1e-12) pentru a crea hărți de stringență restricționată (RS). Hărțile WGP RS au fost modificate manual prin integrarea cu acoperirea noastră anterioară cu nivel scăzut de PM 11 .
În pasul GPM 'assemblyRun' pentru a crea secvențe de secvențe bazate pe BAC, parametrii impliciți pentru îmbinarea celor două secvențe BAC au fost 'minOverlapSeqToSeq = 1000 bp' și 'identitySeqToSeq = 99%', cu suprapuneri necesare la sfârșitul ambelor secvențe. Am folosit Nipponbare RefSeq 17 ca referință pentru a atribui numere de cromozomi asamblării contig, precum și pentru a le ordona și orienta. În plus, doar o copie a secvenței de suprapunere redundante a fost păstrată în contigul asamblat, fără a se acorda preferința determinării piesei din secvența BAC care a fost păstrată. Cu toate acestea, secvențele fără goluri aveau prioritate mai mare decât golurile. Toate contigurile au fost verificate și ajustate manual, după cum este necesar, utilizând browserul GPM 16. Când am folosit contigurile Illumina asamblate pentru a umple golurile dintre două contiguri bazate pe BAC, am selectat numai contigurile Illumin care ar putea interconecta complet două contiguri adiacente bazate pe BAC și, important, astfel de suprapuneri ('minOverlapSeqToSeq = 1000 bp' a 'identitate 99% ") trebuie să apară la sfârșitul ambelor contiguri." Când s-au găsit situsuri redundante în aceste regiuni, secvențele secvențiale bazate pe BAC au fost întotdeauna stocate în ansamblurile finale ale genomului.
Acest document este prima ediție a datelor inițiale pentru asamblarea genomurilor de orez indica ZS97 și MH63 și oferă, de asemenea, primele versiuni ale a două seturi de pseudomolecule de înaltă calitate în comunitatea științifică. Tehnologiile de secvențiere a ADN-ului și programele de asamblare a secvențelor se schimbă rapid, iar seturile de date prezentate aici conțin mai multe tipuri de secvențiere care pot fi utilizate pentru a dezvolta noi metodologii și instrumente software ca intrări de testare.
Mai multe detalii
Cum se citează acest articol: Zhang, J. și colab. Generarea a doi genomi de referință ai orezului indica cu citire lungă PacBio și date de secvențiere Illumina. Știință. Date 3: 160076 doi: 10.1038/sdata.2016.76 (2016).
Citări de date
GenBank KG737749 - KG771717 (2013)
GenBank KG702200 - KG737748 (2013)
Arhivă pentru citirea NCBI SRP071597 (2016)
NCBI Sequence read archive SRP071598 (2016)
NCBI Sequence read archive SRP071944 (2016)
- Îndrăgostit de Tomáš Klus, a știut după două săptămâni că își dorește un copil cu Tamara
- Încetarea pierderii biodiversității și crearea unui sistem alimentar sănătos și durabil în Europa
- Wellness în hotelul Fontana din Brno cu demipensiune pentru doi și un copil cu vârsta de până la 6 ani gratuit, Republica Cehă - Brno
- Yelp folosește educația automată pentru a crea o listă de alimente preferate
- Pot fi patru dintre cele două crime