obiecte
abstract
Materiale și metode
Declarație etică
Toate lucrările la animale descrise în acest studiu au fost aprobate de Comitetul instituțional pentru îngrijirea și utilizarea animalelor (IACUC) de la Universitatea din Arkansas și toate experimentele au fost efectuate în conformitate cu liniile directoare și reglementările aprobate.
Proiectarea unui experiment cu animale
Colectarea sângelui și extracția ADN-ului
Probele de sânge au fost prelevate de la 5 păsări aparent sănătoase pe stilou în zilele 14, 41 și 49 pentru analiza microorganismului pentru a obține 240 de probe de sânge (5 păsări pe stilou × 16 pixuri pe vârsta de prelevare x 3 categorii de vârstă de prelevare). Alte 12 probe de sânge au fost prelevate de la păsări care au suferit BCO (n = 12; 5 și 7 probe de la 41 și 49 de zile, respectiv), permițând analizarea unui total de 252 de probe pentru profilarea ARNr 16S. Probele de sânge au fost prelevate aseptic din vena aripii folosind EDTA Vacutainers. Un ml de probă de sânge a fost centrifugat (5.000 rpm timp de 5 minute la temperatura camerei) folosind o microcentrifugă și 200 μl de strat de tampon a fost colectat într-o cameră sterilă de flux laminar și depozitat la -20 ° C. ADN genomic a fost extras din straturile de tampon. folosind BiOstic® Bacteremia ADN Kit de izolare (MoBio) conform instrucțiunilor producătorului. Probele de ADN au fost analizate folosind un fluorometru Qubit 2.0 (Life Technologies) pentru cantitate și puritate și stocate la -20 ° C.
Protocol PCR pentru amplificarea genei ARNr 16S
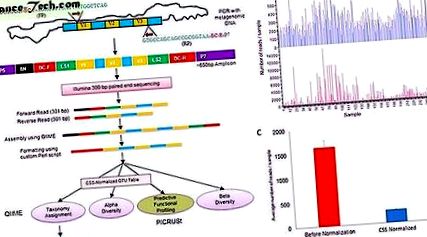
( A ) Diagrama de flux reprezentând pregătirea bibliotecii, formatarea citirilor acceptabile pentru analiza QIIME, care au fost analizate în continuare folosind LefSe și PICRUSt. * Pentru analiza PICRUST s-a utilizat un tabel OTU standardizat. (FP = primer direct, RP = primer invers, V = regiune variabilă a genei ARN 16s, P5 și P7 = primer de secvență Illumina, N = nucleotidă aleatorie, BC-F = cod de bare primer primer, BC-R = cod de bare primer invers) și LS = secvența linker). ( b ) Grafic cu bare care prezintă distribuția a 252 de probe înainte (roșu) și după normalizare (albastru) a tabelului OTU. c ) Grafic cu bare care arată numărul mediu de citiri pe eșantion cu eroare standard înainte (roșu) și după normalizare (albastru).
Imagine la dimensiune completă
Analiza datelor
O diagramă de flux care descrie analiza secvenței ARNr 16S este prezentată în FIG. 1a. Citirea înainte și înapoi a capătului asociat din secvențierea Illumin a fost compilată utilizând informații cantitative asupra ecologiei microbiene, QIIME 1.9.1 25, prin intermediul scriptului join_paired_ends.py folosind metoda fastq-join. Apoi, secvențele codului de bare de citire înainte (BC-F) și citire înapoi (BC-R) au fost unite și unite (capătul 5 ') la secvența asamblată (capătul 3') după îndepărtarea celor 8 secvențe aleatorii de nucleotide și a 27F și 533R secvențe primare după propriul script Perl, care a produs un fișier fastq compatibil cu analiza QIIME ulterioară.
Citirile au fost grupate folosind UCLUST 26 și unitățile taxonomice operaționale (OTU) cu posibilitatea închiderii referinței au fost efectuate folosind ediția 13_8 a bazei de date GreenGenes. Tabelul OTU BIOM (Matricea de observare biologică) a fost normalizat la QIIME (normalize_table.py) folosind metoda Cumulative Scale Sum (CSS), care a fost apoi utilizată pentru atribuții taxonomice, estimarea diversității alfa, identificarea biomarkerului și predicția conținutului metagenom funcțional. Indicii de diversitate alfa au fost comparați utilizând analiza unică a varianței (ANOVA), iar analiza post hoc a fost realizată prin metoda HSD Tukey-Kramer. Analiza similarității (ANOSIM) între grupurile de metadate a fost efectuată folosind metrica de distanță UniFrac neponderată cu QIIME (compare_categories.py). În plus, s-au efectuat analize de coordonare master (PCoA) pentru a estima diversitatea beta cu QIIME (24) folosind metrici UniFrac ponderate și neponderate la diferite adâncimi de eșantionare.
Identificarea biomarkerului a fost efectuată utilizând magnitudinea efectului analizei discriminante liniare (LEfSe) 27. Conținutul funcțional metagenic al microbiomilor din sânge de pui a fost prezis cu PICRUSt (cercetare filogenetică a comunităților prin reconstrucția stărilor neobservate) folosind secvențe genice 16S rRNA 28. Rețeaua bacteriană a fost vizualizată folosind Cytoscape 3.2.1.
Rezultatul
Demultiplexarea QIIME și filtrarea de calitate au produs 4, 153, 965 secvențe asamblate variind de la 40 la 580 bp cu o lungime medie de 469 bp, indicând prezența secvențelor himerice datorită amplificării sau abaterii PCR aberante. În acest studiu, am folosit metoda de referință OTU închisă, deoarece investigăm un nou tip de microbiom pentru care structura comunității, diversitatea și apartenența sunt în mare parte necunoscute. Cu toate acestea, am comparat și rezultatele obținute folosind metoda de referință închisă cu metodele de referință deschise și metodele de picking OTU de novo (tabelul suplimentar 2). Deși referința deschisă și metoda de picking OTU de novo au condus la un număr mai mare de OTU (în medie 196 și respectiv 218), precum și valori mai mari pe eșantion, au fost înregistrate proporții considerabile (în medie 72, 5 și 79, respectiv%). ), care nu au fost incluse în niciun grup taxonomic în baza de date curentă GreenGenes 13_8. În plus față de eliminarea efectivă a tuturor zgomotului sau a secvențelor himerice din analiza ulterioară, am preferat o colecție OTU de referință închisă decât celelalte două metode.
Tabelul de referință închis OTU a avut o adâncime medie a eșantionului de 1.621, pentru fiecare eșantion (± 139, 07), așa cum se arată în FIG. Tabelul OTU a fost normalizat utilizând scalarea cumulativă (CSS) cu QIIME (Fig. 1b, c). Deși valorile medii ale eșantioanelor au fost reduse prin normalizare de peste 6 ori (266, 55 per eșantion), ele au redus și eroarea standard (± 7, 01). Tabelul CSS normalizat OTU a fost utilizat pentru a atribui taxonomie, estimări ale diversității alfa, analiza LEfSe și analiza PICRUt. Analiza diversității beta a fost efectuată la diferite adâncimi de eșantionare și ANOSIM cu permutări de 999 utilizând metrica UniFrac neponderată la aceeași adâncime de eșantionare de 400 de cadre pe eșantion.
Atribuirea taxonomiei
A ) Grafic cu bare la nivelul trunchiului cu diametrul. Diferite litere deasupra fiecărei coloane indică diferențe semnificative (ANOVA, testul Tukey-Kramer, p. 4 .
Categorie experimentală gard gard cu telecomandă. Câmpurile arată percentilele mijlocii, 25 și 75 cu valori în afara extremelor inferioare și superioare. Cutiile cu litere diferite sunt semnificativ diferite (pag
A ) Grafic PCoA de pui sănătoși (n = 171) vs BCO (n = 9) folosind metrici UniFrac ponderate la aceeași adâncime de eșantionare de 400 citite pe probă. ( b ) Parcela PCoA de 14 zile (n = 30), 41 de zile (n = 41) vs 49 de zile (n = 36) pui bătrâni cu metrică UniFrac ponderată la aceeași adâncime de eșantionare de 1 000 de cadre pe probă. ( c ) Diagrama PCoA a starterului (n = 67) vs finisher (n = 154) pui alimentați cu diete folosind metrica UniFrac neponderată la aceeași adâncime de eșantionare de 200 de cadre pe probă.
Imagine la dimensiune completă
O diferență semnificativă în comunitățile bacteriene de pui sănătoși și BCO a fost, de asemenea, ilustrată prin gruparea ierarhică. Puii BCO cu pui de 49 de zile au fost separați de puii sănătoși. Cu toate acestea, puii BCO de 41 de zile nu au prezentat niciun model de grupare semnificativă (Fig. 5a). Analiza rețelei între pui (252 de probe) și OTU (specii bacteriene) a arătat un anumit grad de rezoluție în modelele de interacțiune dintre BCO și pui sănătoși (Fig. 5b), sugerând diferite comunități bacteriene.
A ) Gruparea ierarhică a puilor sănătoși și BCO pe bază de sânge microbian. Arborele filogenetic a fost creat folosind software-ul FigTree V1.3.1 cu metrici ponderate UniFrac folosind date de pre-normalizare. Liniile verzi și roșii reprezintă puii BCO pentru 41 și 49 de zile. Liniile negre sunt pui sănătoși. Numărul de la sfârșitul liniei arată ID-ul probei de pui. b ) Rețeaua bacteriană de microorganisme din sânge de pui produse cu Cytoscape V3.2.1. Nodurile roșii, albastre și albe reprezintă găinile BCO, găinile sănătoase și OTU. Limita verde este rețeaua de pui BCO, iar marginea portocalie este la puii sănătoși.
Imagine la dimensiune completă
Am observat că toate păsările BCO au fost doar din două grupe de vârstă (ziua 41 și 49) și din două grupuri (W35-56 și W1-56). Prin urmare, am efectuat o analiză suplimentară a diversității beta folosind un subgrup de 77 de probe aparținând acestor grupe de vârstă și grupurilor pe bază de buze, inclusiv 65 de păsări sănătoase și 12 păsări BCO. Analiza ANOSIM bazată pe metrici ponderate UniFrac a arătat o separare mai mare între BCO și puii sănătoși (R = 0,5293, p = 0,001) comparativ cu setul total de date (n = 252). Graficul PCoA prezentat în FIG. 6 susține, de asemenea, rezultatul analizei ANOSIM.
Grafic PCoA de pui sănătoși (n = 51) vs BCO (n = 9) folosind metrici UniFrac ponderate la aceeași adâncime de eșantionare de 400 citite pe probă.
Imagine la dimensiune completă
Biomarcatori BCO
Grupurile taxonomice care sunt diferite în abundență între puii sănătoși și BCO au fost identificați utilizând magnitudinea efectului analizei discriminante liniare (LEfSe) cu = 0,05, scor LDA de cel puțin 2 și un număr relativ peste 0,1. Un total de 26 de funcții au avut cantități semnificativ diferite între puii sănătoși și BCO. La nivel de gen, microbiodele de sânge BCO ale găinilor au fost îmbogățite diferențial cu Staphylococcus, Granulicatella și Microbacterium, în timp ce găinile sănătoase au fost îmbogățite cu Pseudomonas, Enhydrobacter și Aquabacterium (Fig. 7b). De asemenea, am constatat că tulpina Firmicutes a fost îmbogățită cu găini BCO. În mod similar, Alphaproteobacteria a fost extrem de îmbogățită în puii BCO, în timp ce Betaproteobacteria și Gammaproteobacteria la puii sănătoși la nivel de clasă (Fig. 7b).
A ) Cladogramă taxonomică realizată din analiza LEfSe. Roșu și verde conțin taxoni îmbogățiți cu BCO și pui sănătoși. Luminozitatea este proporțională cu cantitatea de taxon. ( b ) Impozitele îmbogățite cu pui BCO sunt afișate în roșu cu un scor LDA negativ și puii sănătoși în stare verde cu un scor LDA pozitiv (> 3, 5 în ambele cazuri). Taxonul dintre cele două sublinieri este propus ca numele bazei de date GreenGene.
Imagine la dimensiune completă
Presupusă capacitate genetică funcțională a microorganismelor din sângele de pui
( A ) Numărul relativ de niveluri de KEGG COG la nivelul 1 în microbiomii din sânge de pui. ( b ) Abundența relativă (≥ 0, 5) a căii KEGG la nivelul 3 al căilor funcționale create de software-ul JMP. ( c ) caractere diferențiate (categoria KEGG COG, abundență relativă ≥ 0, 5) produse folosind LEfSe cu scor LDA ≥ 2, 8.
Imagine la dimensiune completă
discuţie
Acesta este primul studiu cuprinzător care analizează microbii bacterieni care există în sângele vertebratelor neumane. În ultimii ani, a existat un interes tot mai mare în caracterizarea microbionului asociat cu diferite țesuturi ale corpului în condiții de sănătate diferite. Părțile corpului care au fost considerate sterile, cum ar fi sângele 30, stomacul 31, vezica urinară 32, plămânii 33, oasele, articulațiile 4 și sânii 34, au microorganismul lor original. Disbioza microbiozei tisulare a fost asociată cu o varietate de boli, inclusiv boli cardiovasculare 10, diabet 35, boală hepatică grasă nealcoolică (NASHD) 36, boală inflamatorie intestinală (IBD) 37, psoriazis 38, obezitate 39, astm cu debut în copilărie 39, boala intestinală funcțională 40 și cancerul colorectal 41. Aici, am examinat microbioza sângelui de pui pentru a identifica potențialii biomarkeri bacterieni asociați cu BCO.
Rezultatul analizei datelor din acest studiu a arătat existența unor comunități bacteriene formate din 30 până la 40 OTU în sângele puilor de pui, indiferent de vârstă și alte condiții de mediu sau de gazdă. Analiza diversității beta (Fig. 4a și 6), analiza ierarhică a clusterului (Fig. 5a) și analiza rețelei bacteriene (Fig. 5b) bazată pe sângele microbian și gruparea ierarhică bazată pe metagenomul previzibil al microbionului sanguin (Fig. 8b) sugerează că comunitățile bacteriene în sângele păsărilor BCO sunt diferite de comunitățile din păsările sănătoase, indicând prezența anumitor presiuni selective care contribuie la schimbarea microbiomerilor din sânge la păsările BCO. Cu toate acestea, doar întregul arbore PD a prezentat o diferență semnificativă în diversitatea alfa între BCO și păsări sănătoase, în concordanță cu microbioza sanguină îndepărtată filogenetic la păsările BCO, după cum se arată în analiza diversității beta (Figurile 4a și 6).
În studiul nostru neînsoțit, probele de sânge de la aceeași turmă au fost direct inoculate pe medii bogate în agar. Rezultatul a arătat că numărul coloniilor din ziua 49 a fost în mod constant mai mare pe pardoselile din sârmă (L35W și W56) în comparație cu așternuturile (L56), sugerând că tensiunea pe pardoseala din sârmă a favorizat bacteremia probabil prin translocație bacteriană prin epiteliul intestinal. În plus, numărul coloniilor din ziua 49 a fost, de asemenea, în mod constant mai mare la păsările șchioapate comparativ cu păsările sănătoase. Aceste rezultate indică o corelație puternică între stresul pardoselii firului, severitatea bacteriemiei și șchiopătarea BCO.
O aplicație practică a acestui studiu a fost identificarea biomarkerilor bacterieni care ar putea fi utilizați pentru identificarea puilor de pui broiler individuali la o vârstă mai timpurie, care sunt susceptibili la dezvoltarea BCO la o vârstă ulterioară. Într-adevăr, analiza datelor din acest studiu a identificat grupuri taxonomice la diferite niveluri care se îmbogățesc semnificativ la păsările BCO în comparație cu păsările sănătoase. Interesant este faptul că genul Staphylococcus este una dintre cele 18 trăsături (inclusiv 3 genuri) îmbogățite semnificativ în probe de BCO, subliniind importanța acestui gen, care a fost adesea izolat de leziunile BCO 3, 4, 5. Recent, Al-Rubaye și colab. 44 a declarat că provocarea broilerilor cu S. agnetis, care a fost cea mai frecvent izolată specie stafilocică din leziunile BCO în studiul său, a crescut semnificativ prejudiciul de la 10 (martor) la 40%, în timp ce provocarea cu un alt izolat Enterococcus faecalis a redus incidența tulburărilor musculo-scheletice Aceasta poate indica importanța potențială a S. agnetis ca agent patogen al BCO la puii broiler, deși datele noastre de profilare a genei 16S rRNA nu au furnizat informații semnificative despre speciile Staphylococcus din cauza rezoluției limitate de atribuire taxonomică.
Cu toate acestea, în acest studiu, BCO a fost detectat doar la păsările cu vârsta de 41 și 49 de zile și, prin urmare, biomarkerii bacterieni au o valoare limitată pentru diagnosticul precoce al păsărilor susceptibile la BCO. Acest aspect ar trebui analizat cu atenție într-un proiect experimental pentru studii viitoare, pentru a permite identificarea potențialilor biomarkeri bacterieni în probele de sânge de păsări tinere care prezic dezvoltarea BCO la grupele de vârstă mai în vârstă.
Microbii de sânge analizați în acest studiu au un efect semnificativ asupra stării de sănătate a puilor de pui broiler, inclusiv a patogenezei BCO, așa cum s-a demonstrat în acest studiu, precum și a altor boli sau condiții de stres la puii broiler.
Mai multe detalii
Cum se citează acest articol: Mandal, RK și colab. Examinarea sângelui microbian și asocierea sa potențială cu condronecroza bacteriană cu osteomielită (BCO) la puii de carne. Știință. reprezentant. 6, 25882; doi: 10.1038/srep25882 (2016).